甜瓜(Cucumis meloL.)是重要的园艺作物,种质资源丰富,分为2个亚种(C. melossp.melo、C. melossp.agrestis)和16个园艺学类群。尽管已有多个甜瓜基因组发表,但基因组组装不完整和野生甜瓜基因组资源的缺乏,仍然阻碍着甜瓜抗性基因的发掘和利用。因此,组装完整的野生甜瓜基因组对解析甜瓜抗病性和广泛适应性的遗传基础至关重要。
近日,中心科研人员与华中农业大学在 the plant journal 在线发表了题为“Complete genome assembly provides a high-quality skeleton for pan-NLRome construction in melon”的研究论文,中心张永兵研究员与华中农业大学孔秋生教授为本文共同通讯作者。该研究从头组装了第一个甜瓜T2T和gap-free水平的完整基因组,并首次构建了甜瓜的泛NLR组(pan-NLRome)。该研究不仅为解析甜瓜基因组结构和抗病基因NLR的多样性提供了重要资源,也为甜瓜泛基因组构建提供了高质量的参考骨架。
来自墨西哥的野生甜瓜种质PI511890带有蔓枯病抗性基因Gsb-3,但该基因尚未被克隆。该研究以PI511890为材料进行基因组组装,以期为克隆Gsb-3基因提供高质量的参考基因组。研究首先基于全基因组重测序数据构建了16个甜瓜园艺类群的系统发育树,发现PI511890属于C. melovarchito类群。然后,使用HiFireads和ultra-long ONTreads进行contig组装,再利用Hi-C测序数据进行染色体挂载。在挂载后的染色体序列上可鉴定到24个端粒、12个着丝粒区域和16个gaps。利用longreads和contigs对gaps进行手动填补,最终实现了T2T和gap-free水平的完整基因组的组装。该基因组大小为375.0 Mb,注释到31,556个编码蛋白质的基因。研究人员基于RIL群体的重测序数据构建了高密度遗传图谱,并通过Hi-C互作热图、BUSCO完整性评估、QV值、LAI值、contigN50和碱基准确性来评估组装的基因组质量,结果均表明PI511890基因组具有高度的组装正确性、碱基准确性、序列完整性和连续性,是目前发表的质量最高和最完整的甜瓜基因组。
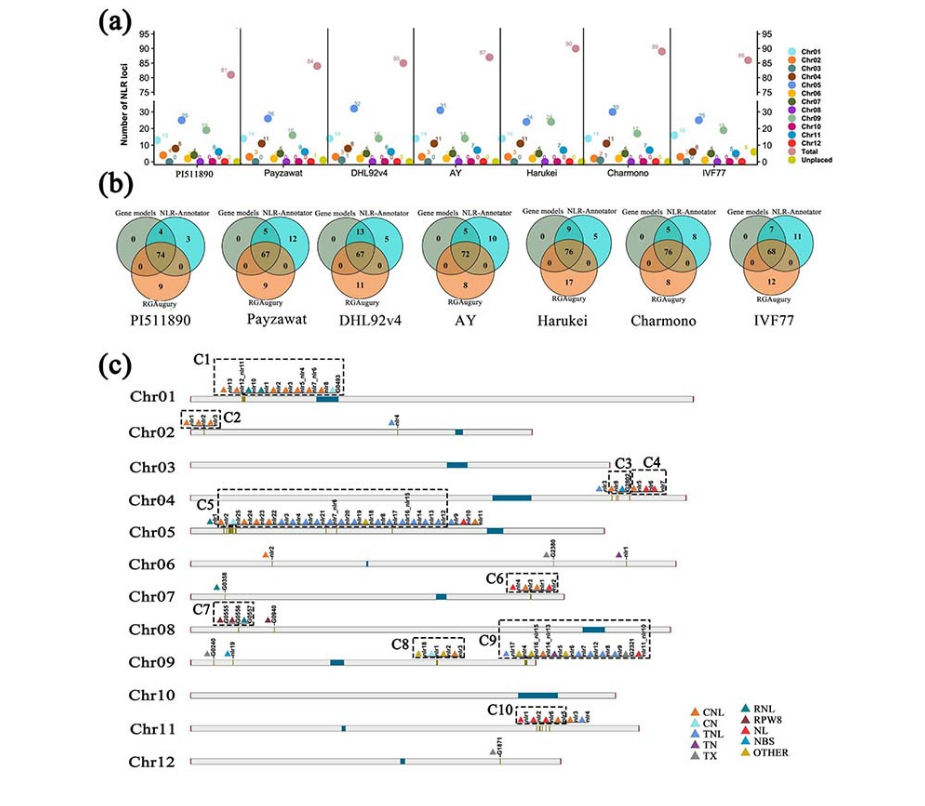
随后,该研究鉴定了甜瓜基因组中的抗性基因类似物(RGA)并对NLR类型的RGA进行重新注释。在PI511890基因组上注释到80个NLR,其中64个NLR成簇分布。该研究进一步对6个已发表的甜瓜基因组的NLR进行了重新注释,构建了一个包含226非冗余NLR的泛NLR组。该泛NLR组包含18个核心NLR基因和208个可变NLR基因。该研究结果不仅为甜瓜遗传改良提供了高质量的基因组资源,同时还为甜瓜抗病基因发掘提供首个可作为无偏参考的泛NLR组。

原文链接:https://doi.org/10.1111/tpj.16705